
外显子组测序是指利用高效的序列捕获技术将基因组中的外显子区域捕获后进行高通量测序的方法。在人类基因中大约有180,000个外显子,虽然外显子组仅占人类基因组的1%(约30MB),但是约85%的致病突变发生在外显子组区域。外显子组测序主要用于识别和研究与疾病、种群进化相关的编码区及UTR区域内的结构变异。结合大量的公共数据库提供的外显子数据,能够更好地解释所得变异结构之间的关联和致病机理。相比传统方法,外显子组测序能够更经济高效的检测外显子区域的变异情况,以此来为临床诊断和个体化用药服务。外显子组测序目前已被广泛的应用于各种癌症、单基因病、复杂疾病和罕见遗传病等研究中。
1、实验方案
捕获平台:Agilent SureSelect Human All Exon V6(最新捕获试剂盒)
测序策略:Illumina NextSeq 500或HiSeq 3000 PE150
数据量:推荐100×以上的深度测序,约10G clean data
2、技术优势
(1)高覆盖度:同等费用下获得更高深度测序,SNP检出率99%;
(2)更高通量:适合大样本量的研究;
(3)经济高效:相比于全基因组重测序,更加经济、高效;
(4)周期短:加快文章发表与加速临床应用。
3、数据分析
3.1 标准信息分析
(1)按标准流程进行数据整理及数据质量评估;
(2)与参考序列进行比对、统计测序深度及覆盖度;
(3)SNP和InDel变异信息检测;
(4)SNP和InDel变异所在基因的功能注释(GO、Pathway);
(5)CNV检测和注释分析;
(6)变异基因保守性预测及致病性分析(SIFT、Polyphen-2、GERP);
3.2 高级信息分析
(1)癌基因/抑癌基因/易感基因筛查;
(2)高频突变基因统计及通路富集分析;
(3)NMF突变特征及突变频率分析;
(4)已知驱动基因筛选;
(5)基因组变异Circos图展示。
4、捕获平台介绍
Agilent SureSelect Human All Exon V5 特点如下:
(1) 捕获范围为50Mb,包括21,522条基因的357,999个外显子区域;
(2) 根据CCDS、RefSeq、GENECODE、miRNABase、TCGA及UCSC数据库设计探针,可有效获得外显子区及其周边区域的变异信息。
(3)周期短,仅需1.5天,测序样品便可准备就绪。
5、技术流程

6、案例分析
案例(1)外显子组测序揭示结直肠癌耐药新机制
该研究通过对129个结直肠癌小鼠进行外显子测序以及拷贝数变异分析,对55个人类肿瘤样本进行目标区域测序,鉴别出了晚期大肠癌中与耐药和药物敏感性相关的6个基因的新突变, ERBB2,EGFR,FGFR1,PDGFRA,MAP2K1和IRS2,并揭示出了对西妥昔单治疗抗产生耐药的新机制。这项研究证实替身可为在人类癌症中评估靶向疗法及将这些反应与潜在基因组学关联起来提供一种系统的途径。在替身中取得成功的疗法或许可以指导个体患者的新疗法及帮助药物开发。
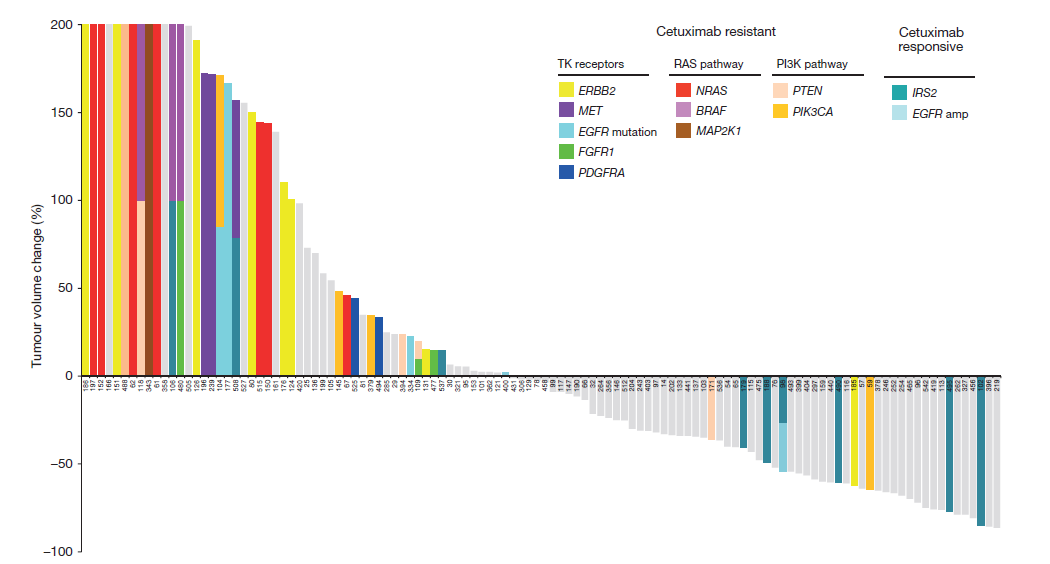
图1 不同体细胞突变对西妥昔单抗治疗肿瘤的增长的影响
案例(2)遗传及化学诱导的KRAS驱动的肺癌基因突变全景图
对非小细胞肺癌模型的小鼠进行外显子测序。KrasLA2诱发的肺癌比致癌物质诱发的肺癌有显著水平的非整倍体以及拷贝数变异(CNV),这表明化学物质诱导的肿瘤与转基因模型拥有不一样的肿瘤发生发展进程。

图1遗传和化学诱导肺癌样本的拷贝数情况
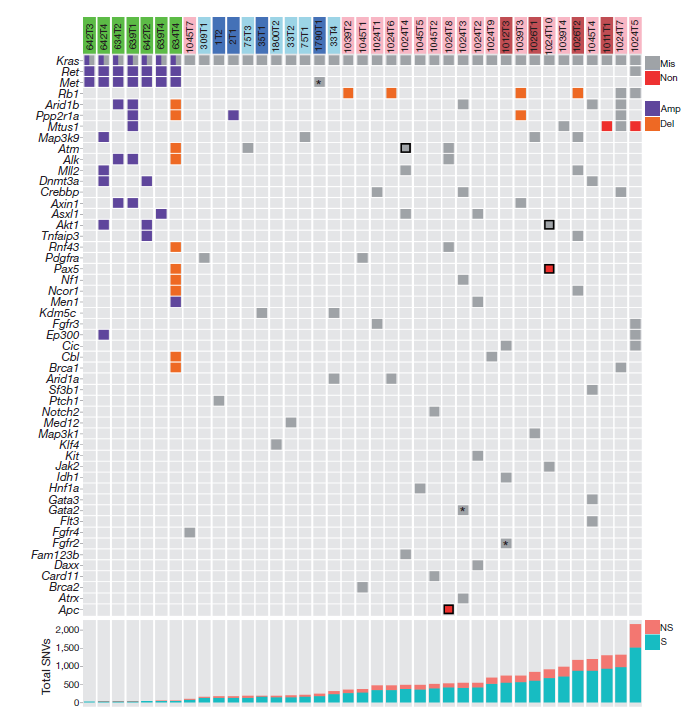
图2 发生在致癌物诱导肿瘤中的驱动SNV
案例(3)外显子组测序发现皮肤基底细胞癌的新的驱动基因和发展路经
皮肤基底细胞癌(BCC)是最常见的一种癌症,90%的人都存在发病风险,尤其是年龄大和紫外线照射多的人。研究人员测序了236名患者的293个皮肤基底细胞癌(BCC)的肿瘤样本,并将测序结果与患者健康细胞的遗传学图谱进行比较,发现了MYCN、PTPN14和LATS1都是BCC的驱动基因。进一步研究表明,这些癌症驱动子也存在于Gorlin综合征中,这可能就是Gorlin综合征患者更容易患BCC的原因。

图1 BCC的突变全景图
参考文献
[1] Bertotti et al. The genomic landscape of response to EGFR blockade in colorectal cancer. Nature, 2015.
[2] Westcott et al. The mutational landscapes of genetic and chemical models of Kras-driven lung cancer. Nature, 2015.
[3] Bonilla et al. Genomic analysis identifies new drivers and progression pathways in skin basal cell carcinoma. Nat Genet, 2016.