
DNA甲基化研究一直是疾病研究的热点,与基因表达、表型性状息息相关。是基因调控的手段之一,通过对位于启动子及基因区CpG岛的甲基化而抑制基因的表达。在维持细胞正常功能、传递基因组印记,胚胎发育、肿瘤发生等方面起着至关重要的作用。
RRBS(Reduced Representation Bisulfite Sequencing)是一种准确且高性价比的DNA甲基化研究方法,在大规模临床样本的研究中有着广泛的应用。RRBS通过限制性内切酶对基因组进行酶切,富集启动子及CpG岛区域,再进行Bisulfite转化, 结合Illumina高通量平台测序,是一种高性价比测序方法。用较小的数据量得到包含最多CpG位点甲基化信息的单碱基精度的甲基化图谱。
应用方向
1. 队列研究、疾病分子分型、临床样本的甲基化 Biomarker 筛选; 复杂疾病及肿瘤发病机制等甲基化研究;
2. 模式动物发育和疾病甲基化研究;
3. 动物遗传育种及进化发育等甲基化研究;
技术优势
1. Bisulfite 结合 NGS 测序, 单碱基分辨率, 最高性价比,金标准方案; 富集 CpG 岛和启动子区域等重要区域,测序目标性强,增加测序深度;
2. RRBS 可以检测高达 6-9M 以上人基因组 CpG 位点; 广泛适合于人、哺乳动物、其他动物及鱼类的物种;
3. 先进的测序平台,采用DNBSEQ-T7测序平台,结果更可靠
4. 单碱基分辨率,做到甲基化位点的定性和相对定量分析
技术流程

图1 RRBS实验流程
送样建议
样本来源:人、大鼠和小鼠(其他样本类型请咨询)
动物新鲜冷冻组织样本1-2g
新鲜或冻存细胞系样本≥10^7个cell
基因组DNA:总量≥ 6ug; 浓度≥ 50ng/ul; 完整性好
备注:不适合 FFPE、cfDNA 等片段化样本
结果展示

图2 甲基化C位点比例分布图
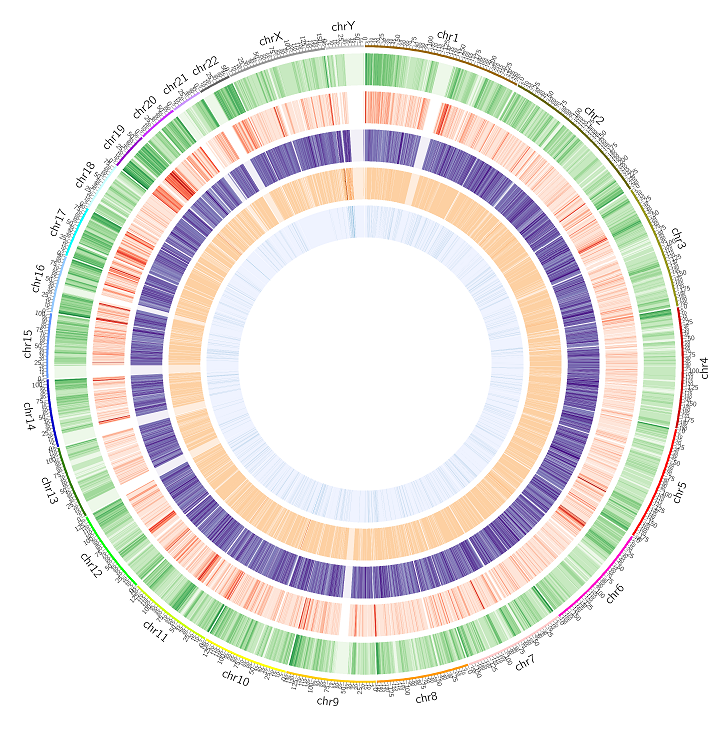
图3 染色体水平甲基化分布
(将全基因组按照500kb的长度划分成了若干窗口,分别统计了每个窗口中的GC含量, gene密度(即基因的数目), CG甲基化水平, CHG甲基化水平, CHH甲基化水平,用于circos绘制。整个图中,除了最外圈染色体核型的标签之外,从外到内的5个heatmap色条,分别表示的是:GC含量(green), gene密度(red), CG甲基化水平(purple), CHG甲基化水平(orange), CHH甲基化水平(light blue)。其中颜色越深表示,密度或者水平越高。)

图4 DMR在锚定区域数量分布比例图(以CG-DMR为例)
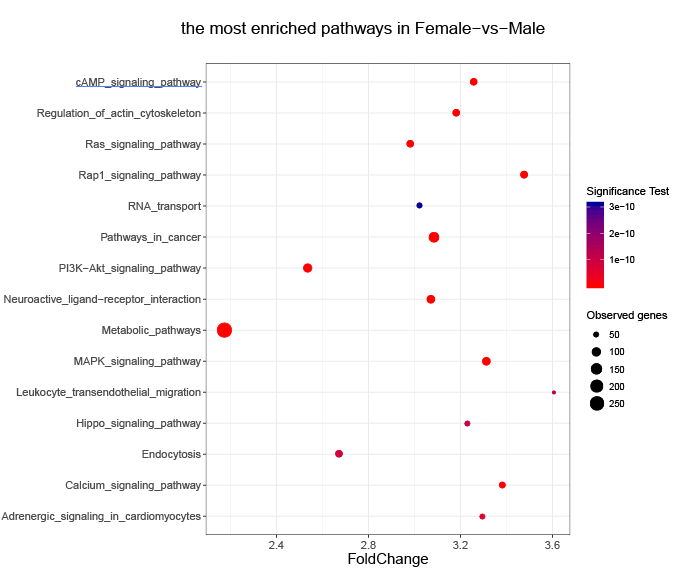
图5 富集的KEGG通路的散点图(以比较组Female-vs-Male为例)
(纵轴表示pathway名称,横轴表示fold change,点的大小表示此pathway中DMR相关基因个数多少,而点的颜色对应于不同的Qvalue范围)
案例分析 :癌前病变到浸润性肺腺癌 DNA 甲基组的演变
Evolution of DNA methylome from precancerous lesions to invasive lung adenocarcinomas[1].

研究方案:在肺腺癌的早期癌变过程中 DNA 甲基化谱和甲基化肿瘤内异质性(ITH)的演变尚未得到系统的研究; 本课题通过 RRBS 方案对 124 个手术切除样本(14 个非典型腺瘤性增生AAH; 15 个原位腺癌 AIS; 22 个微侵袭性腺癌 MIA 和 11 个侵袭性腺癌 ADC 及病灶旁样本)进行单碱基分辨率的甲基化测序。
研究结果:1) 在晚期病变中甲基化变异逐渐增加,甲基化水平明显升高; 2) 从甲基化变异推断的系统发育模式类似于基于体细胞突变的模式,表明 DNA 甲基化和体细胞突变的演化是平行的; 3) 在晚期疾病中 T 调节细胞与 CD8+T 细胞的比率较高,这意味着随着肿瘤进展免疫抑 制;4) 全局低甲基化增加与更高的突变负担、拷贝数变异负担和更高的 Treg/CD8 比率相关, 表明 DNA 甲基化在肺腺癌早期癌变过程中对染色体不稳定性、突变和肿瘤免疫微环境的潜在影响。
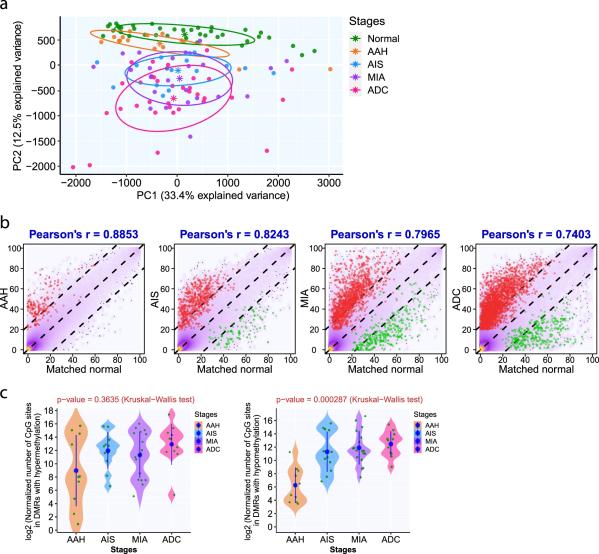
图 6. DNA 甲基化变化随癌前病变的进展而增加
参考文献:Hu, X., et al., Evolution of DNA methylome from precancerous lesions to invasive lung adenocarcinomas. Nat Commun, 2021. 12(1): p. 687.